Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
石田 恒; 郷 信広
no journal, ,
ホリデイ構造は、DNA相同組換えの普遍的なDNA中間体である。大腸菌では、RuvA-RuvB複合体がホリデイ構造の分岐点移動を促進する。RuvAはホリデイ構造特異的DNA結合蛋白質でる。RuvBは2つの6量体がホリデイ分岐DNAと結合し、ATP加水分解エネルギーを用いてホリデイ分岐点の移動反応を触媒する。分岐点移動反応を原子レベルで理解するには、分岐点移動におけるエネルギー地形及び遷移状態を解析する必要がある。そこで、ホリデイ分岐DNAに調和型ポテンシャルを加えることで分岐点移動のエネルギー地形を調べるアンブレラサンプリングシミュレーションを実行した。系は、E.Coli由来のRuvA4量体-ホリデイ分岐DNA(25塩基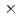 4鎖)複合体の立体構造(PDBコード:1C7Y)を用いた。結果、分岐点移動における自由エネルギー障壁は約15kcal/molと見積もることができた。また自由エネルギー地形とホリデイ分岐DNAの構造変化の関係を解析したところ、分岐点移動開始時には対のない13番目塩基に隣接する12-14番塩基対の水素結合が不安定化することがわかった。塩基対の水素結合が崩れた後、対を作らない塩基と隣の塩基のスタック間距離が大きくなる。遷移中間状態では、対を作らない塩基は分岐点中心にあるRuvA酸性ピンと安定化する。特に、酸性ピンはグリコシド結合角を安定な位置に維持することがわかった。以上により、RuvAがホリデイ分岐DNAの分岐点移動を促進する仕組みを構造的に理解できた。
4鎖)複合体の立体構造(PDBコード:1C7Y)を用いた。結果、分岐点移動における自由エネルギー障壁は約15kcal/molと見積もることができた。また自由エネルギー地形とホリデイ分岐DNAの構造変化の関係を解析したところ、分岐点移動開始時には対のない13番目塩基に隣接する12-14番塩基対の水素結合が不安定化することがわかった。塩基対の水素結合が崩れた後、対を作らない塩基と隣の塩基のスタック間距離が大きくなる。遷移中間状態では、対を作らない塩基は分岐点中心にあるRuvA酸性ピンと安定化する。特に、酸性ピンはグリコシド結合角を安定な位置に維持することがわかった。以上により、RuvAがホリデイ分岐DNAの分岐点移動を促進する仕組みを構造的に理解できた。
河野 秀俊; Kumar, S.*; Ahmad, S.*; Ara zo-Bravo, M. J.*; 藤井 聡*; 郷 信広; 皿井 明倫*
zo-Bravo, M. J.*; 藤井 聡*; 郷 信広; 皿井 明倫*
no journal, ,
転写因子などのDNA結合蛋白質はDNA配列に特異的に結合する。DNA結合蛋白質のDNA配列の認識は、おもに静電相互作用,水素結合,ファンデルワールス相互作用などの塩基とアミノ酸残基の直接的な相互作用によって行われていると考えられてきた。しかし、多くの蛋白質-DNA複合体構造が明らかになるにつれ、直接的な相互作用の数が少ないこと,直接に蛋白質と相互作用をしていない塩基配列を変えても結合強度が変わることなどが明らかになり、直接的でない認識、つまり、間接認識の重要性も認知されるようになってきた。間接認識とは、配列に依存したDNAの特異構造や曲がりやすさなどの構造情報や物性情報を通してDNA配列を蛋白質が間接的に認識することをさす。したがって、蛋白質とDNAの認識機構を解明するには、この両者の寄与を明らかにする必要がある。しかしながら、直接認識と間接認識の寄与を定量化することが難しいため、その寄与バランスはよくわかっていない。われわれは、両者の寄与を定量化する方法を開発し、さまざまな蛋白質-DNA複合体についてその寄与バランス調べた。直接認識は、蛋白質-DNAの複合体構造から抽出されたアミノ酸と塩基の空間分布から計算されるknowledge-based potentialを、間接認識では、複合体のDNA構造の配列依存性から抽出されるknowledge-based potential及びさまざまなDNA配列の分子動力学計算による構造アンサンブルから計算されるpotential of mean forceを用いて、それぞれの寄与をエネルギーとして定量化した。計算したエネルギーは、ランダムDNA配列とのエネルギーの差としてそれぞれで標準化し(Z-score化)、直接認識と間接認識の寄与を評価した。本発表では、約200の蛋白質-DNA複合体における寄与バランスを報告する。
Kim, T. P. O.; 由良 敬; 郷 信広
no journal, ,
蛋白質-RNA複合体は遺伝子発現の機構において数多く現れる。蛋白質とRNAの相互作用面を解析することで、相互作用の統計的な法則を探し、蛋白質とRNAの相互作用面を予測する計算科学的手法を開発した。その後、開発した方法をmRNA核外輸送に関する蛋白質TAP, Mex67などとmRNAとの間の相互作用面に適用した。
松本 淳
no journal, ,
本研究は、数百塩基対程度の長さのDNA断片の低振動数の基準振動を解析するためにわれわれが新たに開発した計算手法を用いて行う。この手法の利点は、比較的長いDNA断片の計算を非常に高速に行いながら、しかも局所構造とその柔軟性の配列依存性を考慮している点である。最初に線形のDNAに対して、この手法は適用され、ローカルな構造変化とグローバルな構造変化の関係が明らかにされた。さらに、環状DNAへの応用では、理論と極めて良い一致を示し、円形DNAから8の字型DNAへの構造変化についての新しい視点を与えた。今回われわれは、より現実に近いモデルを用いて、環状DNAの局所構造の性質とグローバルな立体構造の性質との関係を研究する。われわれは、DNAの環状化実験が既に行われているp53応答配列DNAの異常なやわらかさに注目し、その起源を局所構造の性質から探る。
中川 洋
no journal, ,
非干渉性中性子散乱法はピコからナノ秒スケールのタンパク質のダイナミクスを調べるのに有効な手法の一つである。タンパク質のダイナミクスの特性を理解するために、スタフィロコッカルヌクレアーゼを用いてダイナミクスの水和と温度の効果を非干渉性中性子非弾性散乱により調べた。低温ではタンパク質のボソンピークが低エネルギー領域で観測されたが、水和によってその位置が高振動側へシフトした。このことは低振動モードが水和とカップルしていることを示す。高温でのデバイワーラーファクターの異常増加は、動力学転移とよばれる平均自乗変位の増加に対応する。またこれに伴い準弾性散乱が現れる。準弾性散乱は緩和や確率的な運動のような非調和運動に由来する。水和依存と非依存の動力学転移が観測された。準弾性散乱の解析は転移の動力学的性質を明らかにする。この動的性質はタンパク質の機能に本質的であると考えられる。
中川 洋; 城地 保昌*; 片岡 幹雄*; 郷 信広
no journal, ,
タンパク質の構造が揺らぐとき、その動きに伴いタンパク質の体積は変化する。このようなタンパク質の体積揺らぎと、タンパク質内部に存在するキャビティと呼ばれる空間的な空隙の存在との関係が議論されている。キャビティの体積は外部の圧力に敏感に反応し、例えばタンパク質を加圧するとキャビティの体積は減少し、タンパク質の体積揺らぎが小さくなる。タンパク質の動力学転移は構造の非調和的な運動により生じ、このような運動がタンパク質の機能にかかわっていると考えられている。運動の非調和性は、タンパク質全体にわたる低振動運動の特徴であり、タンパク質の体積揺らぎに関与していると考えられる。したがって、タンパク質のキャビティの存在はタンパク質の動力学転移が生じるメカ二ズムに関係していると考えられる。本研究では、Staphylococcal nucleaseを用いて2000気圧の高圧下での分子動力学計算を行った。タンパク質のキャビティと低振動運動及び動力学転移の圧力効果を調べ、それらの関連性からタンパク質の動力学転移が生じるメカ二ズムを明らかにすることを目的とした。分子動力学計算の結果、高圧下では常圧下に比べキャビティの体積や平均自乗揺らぎが減少することがわかった。また主成分解析からは、常圧下では低振動の運動は幾つかのエネルギー極小間をジャンプするような運動が見られるが、高圧下では比較的少数のエネルギー極小にトラップされる傾向にあった。このことは高圧条件下ではタンパク質の非調和的な運動が抑制されることを示唆する。
樋口 真理子; 石田 恒; 北尾 彰朗*; 山縣 ゆり子*; 郷 信広
no journal, ,
放射線や紫外線あるいは細胞内での代謝活動によって生じる活性酸素はヌクレオチドを酸化させる。酸化したヌクレオチドはDNAに取り込まれると突然変異の原因となる。E. Coliから発見された蛋白質MutTは酸化したヌクレオチド8-oxo-dGTPを加水分解し8-oxo-dGMPとすることでDNAに取り込まれることを防いでいる。MutTは8-oxo-dGMPとdGMPを大きな結合力の差(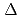
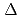 G=6.1kcal/mol)で識別する。しかし、8-oxo-dGMPとdGMPの構造の差は8位の酸素と水素、及び7位の窒素に水素が付加しているか否かでしかない。また、山縣らによりX線結晶構造解析を用いて解かれた8-oxo-dGMP-MutT構造を基質と結合していないMutTの構造と比較すると、おもに基質周りのループ部分が基質を囲むように大きく構造変化していることがわかる。これらのことより、基質によるMutTの構造変化と基質認識に関連があると予想できる。この関連を調べるため、上記の構造をもとに単体のMutT、それぞれの基質との複合体についてAMBER7を用いて分子動力学計算を行い比較した。dGMP-MutTは8oxo-dGMP-MutTの基質をdGMPに入れ替えたものを初期構造とした。その結果、8oxo-dGMP-MutTは基質周りのループが閉じたまま安定で、MutTの構造揺らぎも小さいが、dGMP-MutTでは、MutTの構造が基質と結合していない時の構造に近づき構造揺らぎが大きくなった。また、基質を囲むループが開いたこと、基質結合部位に位置する119Asnの側鎖が回転することにより、基質-MutT間の安定な水素結合の数が減少した。これらはdGMPとMutTの結合時にエントロピー及びエンタルピーの変化が小さいことと定性的に矛盾しない結果である。
G=6.1kcal/mol)で識別する。しかし、8-oxo-dGMPとdGMPの構造の差は8位の酸素と水素、及び7位の窒素に水素が付加しているか否かでしかない。また、山縣らによりX線結晶構造解析を用いて解かれた8-oxo-dGMP-MutT構造を基質と結合していないMutTの構造と比較すると、おもに基質周りのループ部分が基質を囲むように大きく構造変化していることがわかる。これらのことより、基質によるMutTの構造変化と基質認識に関連があると予想できる。この関連を調べるため、上記の構造をもとに単体のMutT、それぞれの基質との複合体についてAMBER7を用いて分子動力学計算を行い比較した。dGMP-MutTは8oxo-dGMP-MutTの基質をdGMPに入れ替えたものを初期構造とした。その結果、8oxo-dGMP-MutTは基質周りのループが閉じたまま安定で、MutTの構造揺らぎも小さいが、dGMP-MutTでは、MutTの構造が基質と結合していない時の構造に近づき構造揺らぎが大きくなった。また、基質を囲むループが開いたこと、基質結合部位に位置する119Asnの側鎖が回転することにより、基質-MutT間の安定な水素結合の数が減少した。これらはdGMPとMutTの結合時にエントロピー及びエンタルピーの変化が小さいことと定性的に矛盾しない結果である。
由良 敬
no journal, ,
生体中で実際に機能しているタンパク質は、非常に大きな複合体(生体超分子)である場合が多い。そのため生命現象を物質に基づいて理解するためには、生体超分子を構成する全原子の座標情報が重要となる。生体超分子の構造は電子顕微鏡で明らかにされる場合が多いが、電子顕微鏡では一般的には解像度が1.2nm程度までしか得られない。原子レベルの構造との間には解像度に10倍程度の差がある。このような状況においてなすべきことは、生体超分子を構成する要素タンパク質の立体構造をX線結晶解析によって明らかにし、各要素分子を電子顕微鏡で得られている全体構造にあてはめ、生体超分子の原子構造を作り出すことであると考えられる。電子顕微鏡で得られる生体超分子の全体構造に、生体超分子を構成する各要素タンパク質の原子構造をあてはめる方法の開発は、世界的にも活発になってきている。それらの研究によって、少なくとも次の二つの問題点が明らかになってきた。(1)X線結晶構造解析で判明している要素タンパク質の立体構造と、生体超分子を構成している時の要素タンパク質の立体構造との間には大きな構造変化が起こっている場合がある。(2)電子顕微鏡像に要素タンパク質の構造をあてはめたときに、あてはめ結果を一意的に決定することができない場合がある。そこで、どのような情報を導入すればこれらの問題を克服できるかを検討するために、立体構造既知の生体超分子と要素タンパク質の組合せが明確になるデータベースを構築し、生体超分子構造と要素タンパク質構造とを比較した。本データベースに基づき、生体超分子形成時に要素タンパク質はどのような構造変化を起こし、要素タンパク質のどのような部位が相互作用部位になっているかを議論する。
藤原 悟; 松本 富美子*; 高橋 伸明
no journal, ,
骨格筋・心筋の収縮制御は細いフィラメント中のトロポニン(Tn)-トロポミオシン(Tm)系により行われている。Ca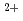 のTnCへの結合から始まるTnI, TnT, Tmの一連のコンフォメーション変化である筋収縮制御の初期過程の分子機構の理解のためには細いフィラメント中でのTnサブユニットのそれぞれの構造を知ることが重要である。われわれは、その第1段階として、重水素化TnC(dTnC)を用いて細いフィラメント中におけるTnCの構造を中性子散乱及び繊維回折法により調べた。今回、TnCに続いてTnIの重水素化を行い、dTnIを含む再構成細いフィラメント(dTnI-RTF)及びnativeな細いフィラメント(NTF)のそれぞれについてCa
のTnCへの結合から始まるTnI, TnT, Tmの一連のコンフォメーション変化である筋収縮制御の初期過程の分子機構の理解のためには細いフィラメント中でのTnサブユニットのそれぞれの構造を知ることが重要である。われわれは、その第1段階として、重水素化TnC(dTnC)を用いて細いフィラメント中におけるTnCの構造を中性子散乱及び繊維回折法により調べた。今回、TnCに続いてTnIの重水素化を行い、dTnIを含む再構成細いフィラメント(dTnI-RTF)及びnativeな細いフィラメント(NTF)のそれぞれについてCa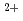 を含む/含まない状態の配向試料を、キャピラリー封入した試料ゾルを18Tの磁場中(理研播磨・前田構造生物化学研)に静置することにより調製し、その中性子繊維回折測定を行った。測定は日本原子力研究開発機構所有の中性子小角散乱装置SANS-Jを用いて行った。得られた回折像から子午線上に観測されるTn由来の反射を抽出し、その3次反射までの振幅を計算した。Ca
を含む/含まない状態の配向試料を、キャピラリー封入した試料ゾルを18Tの磁場中(理研播磨・前田構造生物化学研)に静置することにより調製し、その中性子繊維回折測定を行った。測定は日本原子力研究開発機構所有の中性子小角散乱装置SANS-Jを用いて行った。得られた回折像から子午線上に観測されるTn由来の反射を抽出し、その3次反射までの振幅を計算した。Ca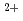 の有無のそれぞれについて、dTnI-RTF及びNTFの振幅の差が観測された。これらの散乱振幅及びdTnCの実験から得られた散乱振幅について、細いフィラメント繊維軸への1次元投影構造モデルを用いた解析を行い、Tn複合体中におけるTnC及びTnIの配列について議論した。
の有無のそれぞれについて、dTnI-RTF及びNTFの振幅の差が観測された。これらの散乱振幅及びdTnCの実験から得られた散乱振幅について、細いフィラメント繊維軸への1次元投影構造モデルを用いた解析を行い、Tn複合体中におけるTnC及びTnIの配列について議論した。